クローン病の予後予測と脂肪組織萎縮の関係
共同第一著者として携わった研究がthe American Journal of Pathologyに受理されました。
テーマは『手術時に取得した切片からクローン病の2年以内の再発を予測するモデルを作成→モデルの出力を解析して再発に関わる因子を調べる』でした。コードはgithubに公開されています。
全文はここをご確認ください。
"Deep learning analysis of histologic images from intestinal specimen reveal "adipocyte shrinkage" and mast cell infiltration to predict post-operative Crohn's disease." Hiroki Kiyokawa, Masatoshi Abe, Takahiro Matsui, Masako Kurashige, Kenji Ohshima, Shinichiro Tahara, Satoshi Nojima, Takayuki Ogino, Yuki Sekido, Tsunekazu Mizushima, Eiichi Morii(https://doi.org/10.1016/j.ajpath.2022.03.006).
クローン病とは
主に若者に多い消化管の至る所に発生しうる慢性炎症性疾患です。
ノイズと背景の多いWSIへの対処
WSIは病理組織プレパラート全体をデジタルスキャンしたものです。
WSIの前処理はkaggleのPANDAコンペなどでも扱われており、背景は白で組織はピンク〜紫なので巨大画像を256*256などのpatchに分割→ほとんど余白であるpatchは捨てる、といった前処理がされます。
今回のデータでは、組織以外の余白部分が多いだけでなく、①スライドガラスの縁があれば黒い線として写ってしまう。②手術で取得したものであるというデータ作成過程上の特徴により組織以外の紫~黒の物体(血液など)が小さいながらも写っている。という2点が前処理上ネックとなりkaggle上の前処理をするだけではうまくいきませんでした。
そこで、一旦元画像上で組織部分だけ2値化→組織以外の部分は白に塗り直す、という処理をあらかじめ行うことにより色の濃いノイズに対応しました。組織のpatch作成はPANDAコンペで紹介されていた組織部分をより効率的に除く処理を参考にしました。

モデル作成
取得したpatchを入力としてWSIについていた再発するかどうかの2値のラベルを予測するモデルを学習させました。モデルはお馴染みの"tf_efficientnet_b5_ns"を使っています。
augmentationにはベンガルコンペで紹介されていたaugmixとgridmaskを使用しました。手元の実験ではcutmixも有効でした。GeM poolingは若干良い程度でavg poolingで十分だった気もします。細胞診の分類でもcutmixが有効だったのでpatch分割とcutmixは相性が良いのではないかと個人的に考えています。
予測の解析
予測値が両極に近いpatchを見てみると漿膜下脂肪組織に差があるのではないかという仮説が生まれました。

そこで脂肪細胞数/細胞の面積/細胞間距離/細胞の丸み(楕円fittingしたときの長軸)/短軸の割合/間質の面積を計測しました。この作業にはインスタンスセグメンテーションが必要だったのですが自分でmaskを作成してモデルを構築だけのパワーがなかったので深層学習は使用せずにopencvなどを駆使しました。
以下が脂肪細胞の解析結果です。
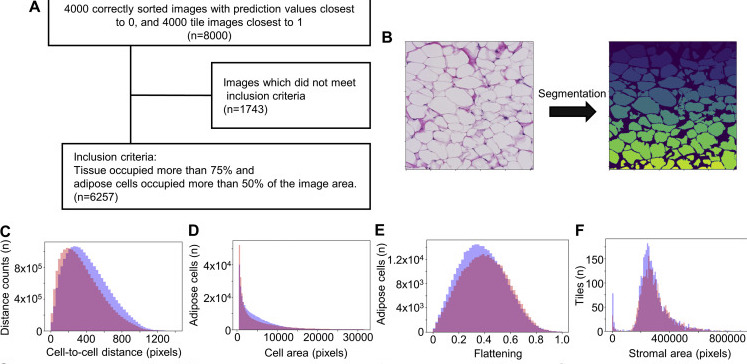
結果として『脂肪細胞の収縮が、疾患の再発に関係する』ことが示唆されました。
感想
解析実際にやっていたのは2020年冬~2021年春でまだkaggleは"見る専"だった頃のものですが見よう見真似でkaggle上から引っ張ってきたアイデアが効いたり、自分なりに前処理を変更したりと楽しかった記憶があります。
引用
この記事の画像が全て元の論文から引用されました。
謝辞
本研究の計算はすべて大阪大学医学部医学科学生用計算機上にて行いました。
WSI関連雑記
WSIをタイル分割してモデル作成→分散表現を得て後段タスクに活用(https://arxiv.org/abs/1901.11112)
自己教師あり学習を用いて表現を得るなどの工夫のしがいがあって楽しそうです。
- 前の記事 : 清麗戦の研究:早く負けるが得か?
- 次の記事 : 非負値行列因子分解(NMF)を用いたグリッドパターンの創発
- 関連記事 :

